Geneious Prime マルチプルアライメント
このチュートリアルでは、後天性免疫不全症候群(AIDS)の原因であるヒト免疫不全ウイルス(HIV)と類人猿の免疫不全ウイルス(SIV)の配列アライメントを作成していきます。
1.データの準備
本チュートリアルで利用する配列は以下の通りです。
- アフリカの霊長類由来 SIV の env 配列
- 飼育下の非アフリカ種(アカゲザル、スタンプオマキザル、ブタオマキザル)に感染した SIV 由来の env 配列
- HIV-1、HIV-2 由来の env 配列
env 遺伝子は全てのレトロウイルスに存在し、ウイルスの表面に位置し、宿主の細胞表面の受容体と相互作用するウイルスエンベロープ糖タンパク質をコードしている遺伝子です。
データを以下よりダウンロードしてください。
ダウンロードしたファイルを Geneious 上にドラッグ&ドロップするか、メニューバーの「File」→「Import」→「Files...」を選択し、ファイルをインポートします。
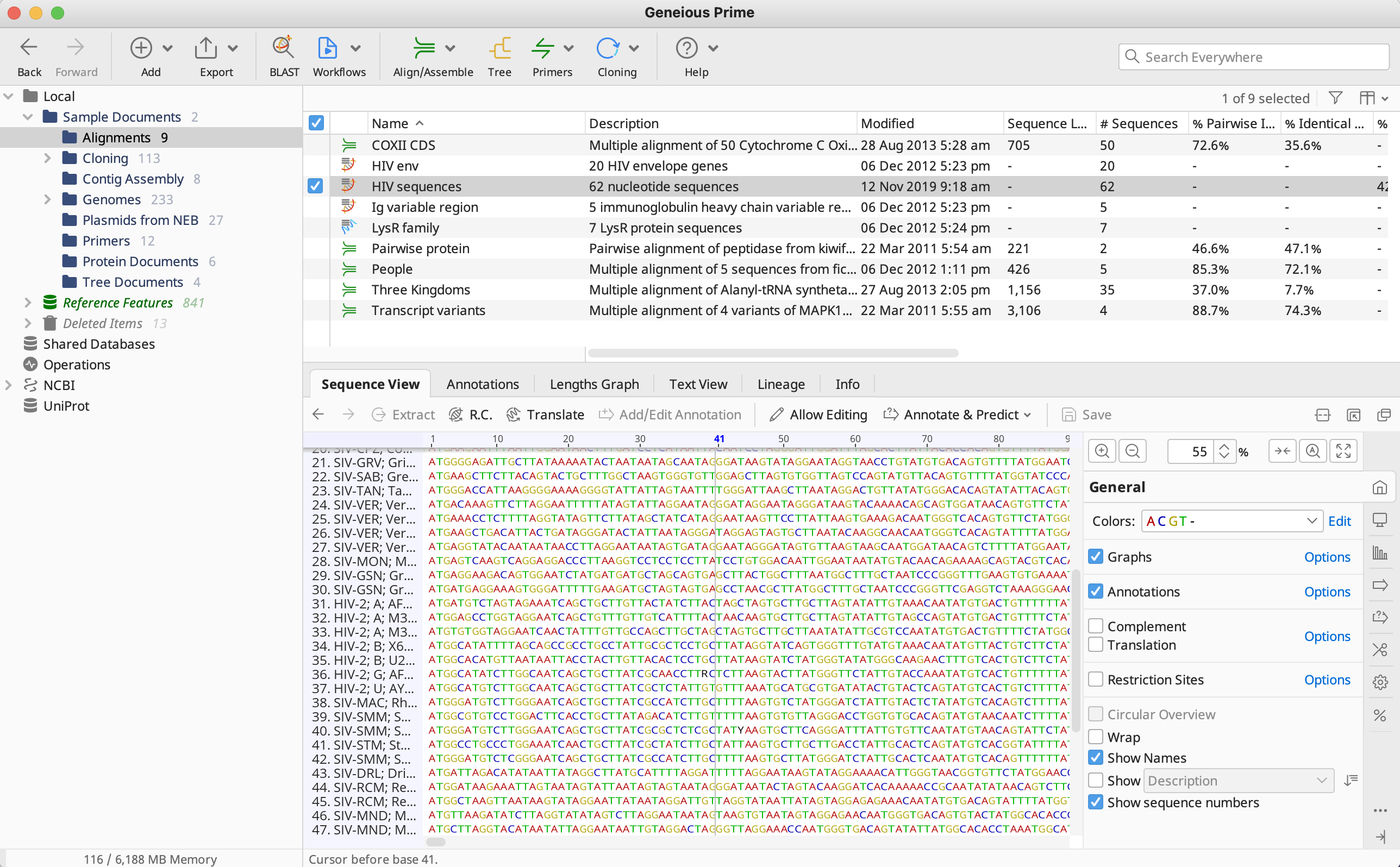
HIV_sequences の中身は、以下のフォーマットで表示されます。
ウイルスの種類・SIVの場合は霊長類の種名・HIV-1とHIV-2の場合はグループ名とサブタイプ・アクセッション番号
例1:HIV-1; B; K03455( HIV-1 のグループ M・サブタイプ B の env 配列)
例2:SIV-SMM; Sooty mangabey; AF334679(スーチーマンガビー由来の SIV の env 配列)
2. 配列アライメントの作成
それでは、全62配列をアラインしていきます。ツールバーの Align/Assemble から Multiple Align へと進みます。
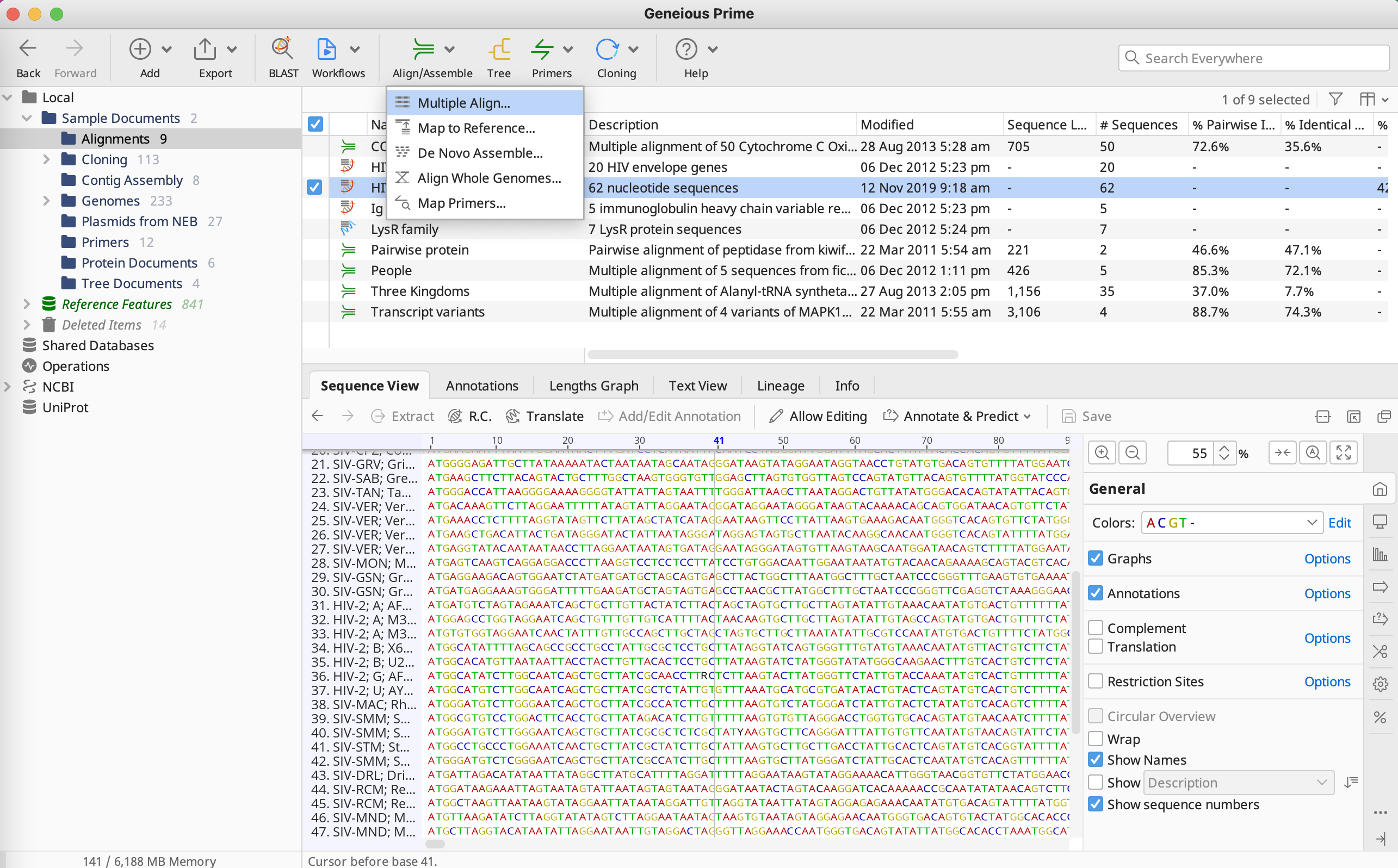
Geneious には以下の3種類のアラインメントプログラムが用意されています。
- Geneious aligner
- MUSCLE
- Clustal Omega
さらに、MAFFT aligner のプラグインも用意されています。これらのアライナーの詳細については、Biomatters社が提供するKnowledge BaseWhich multiple alignment algorithm should I use?をご覧ください。
比較する env 遺伝子の配列は N 末および C 末の相同性が高いので、今回は MUSCLE アライナーを使用します。アライメントオプションから MUSCLE Alignment を選択します。個々のパラメータを設定することもできますが、今回はデフォルトのパラメータを使用します。
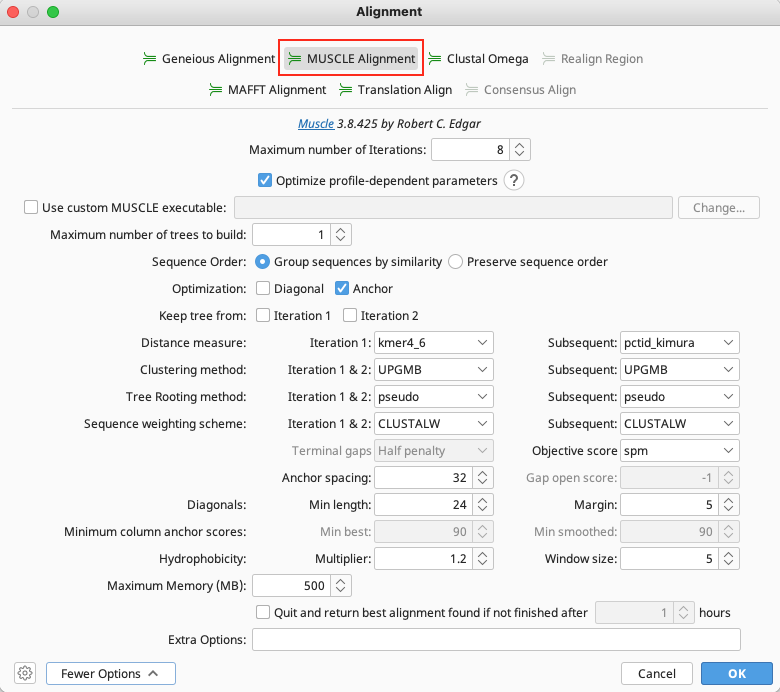
すでにパラメータを変更していて、デフォルトのそれが分からない場合は、ウィンドウの左下にある ![]() をクリックし、Reset to defaults をクリックしてください(当該部分がグレーアウトしている場合は、デフォルトのパラメータがすでに設定されています)。
をクリックし、Reset to defaults をクリックしてください(当該部分がグレーアウトしている場合は、デフォルトのパラメータがすでに設定されています)。
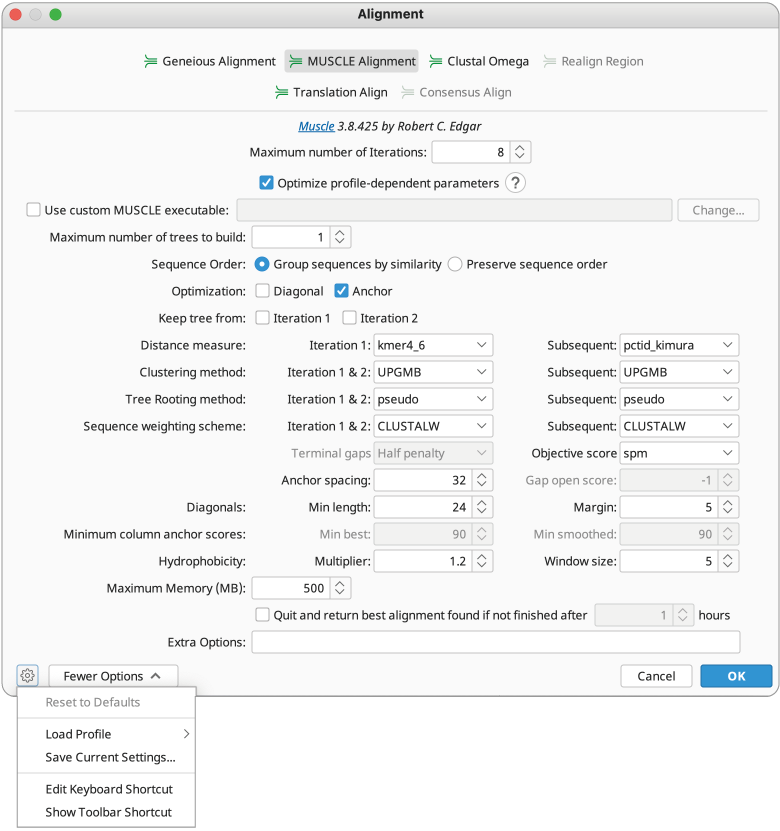
OKボタンをクリックするとアライメントが開始します。
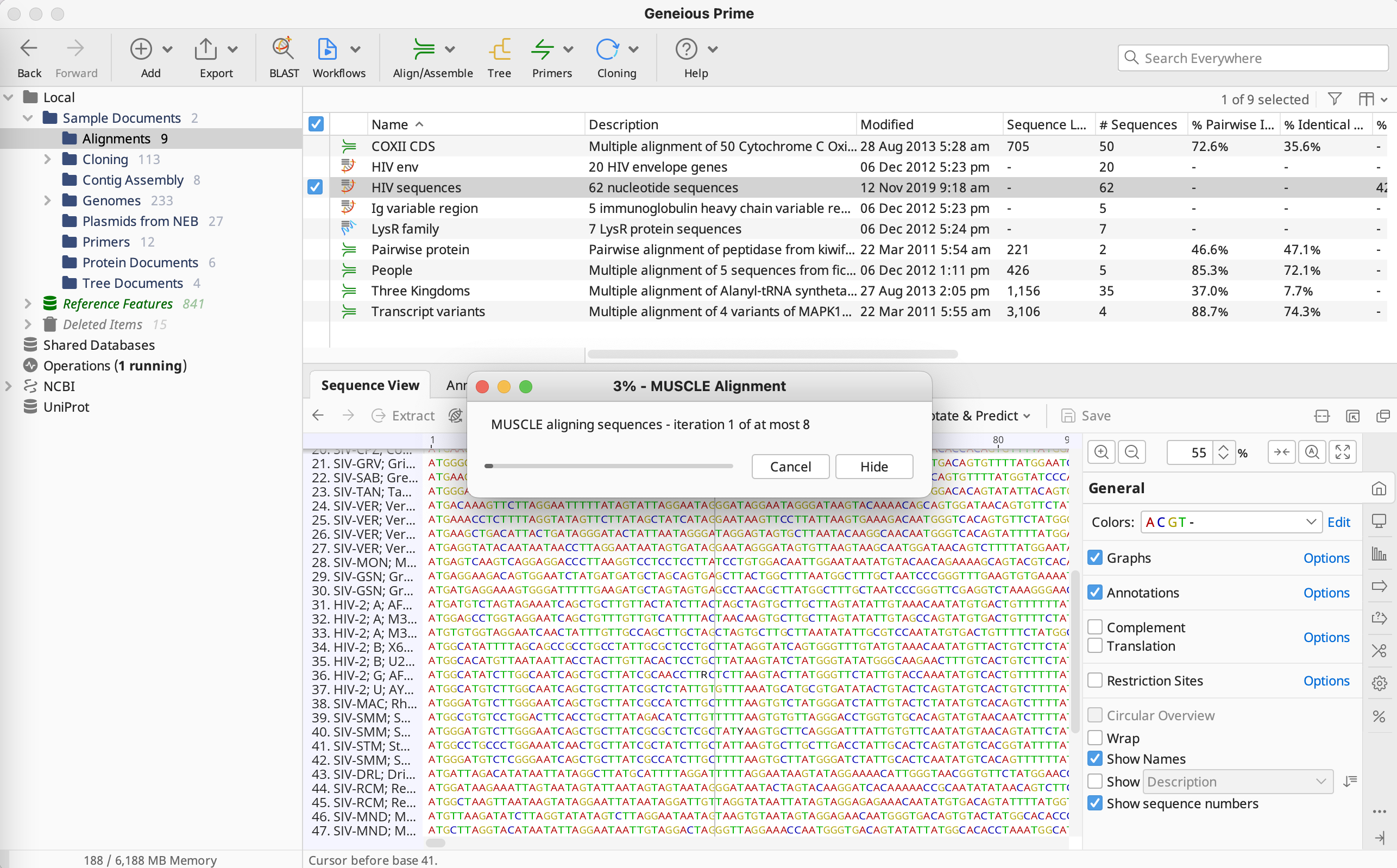
アライメント完了後、パネル上部にある ![]() (Zoom to level機能)を利用して、塩基の表示を拡大します。
(Zoom to level機能)を利用して、塩基の表示を拡大します。
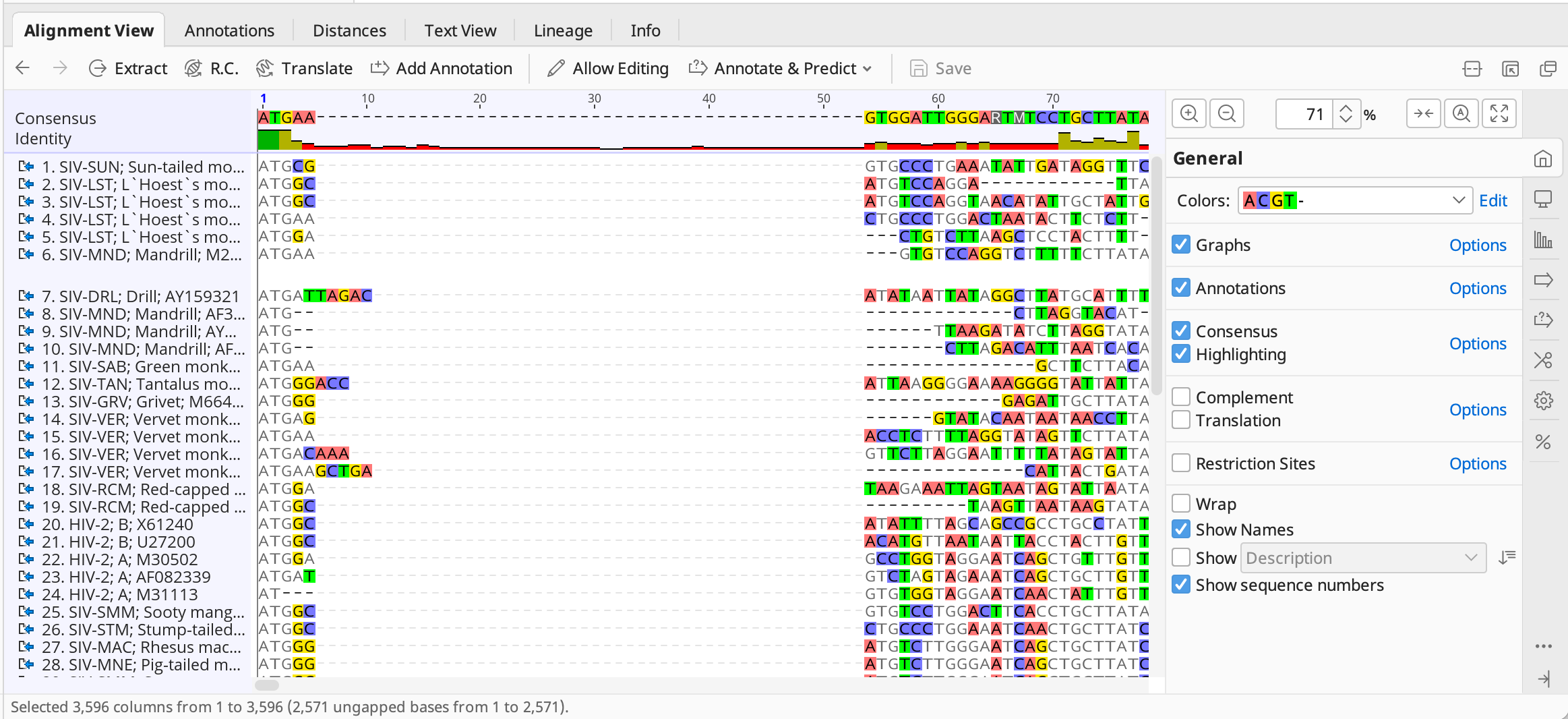
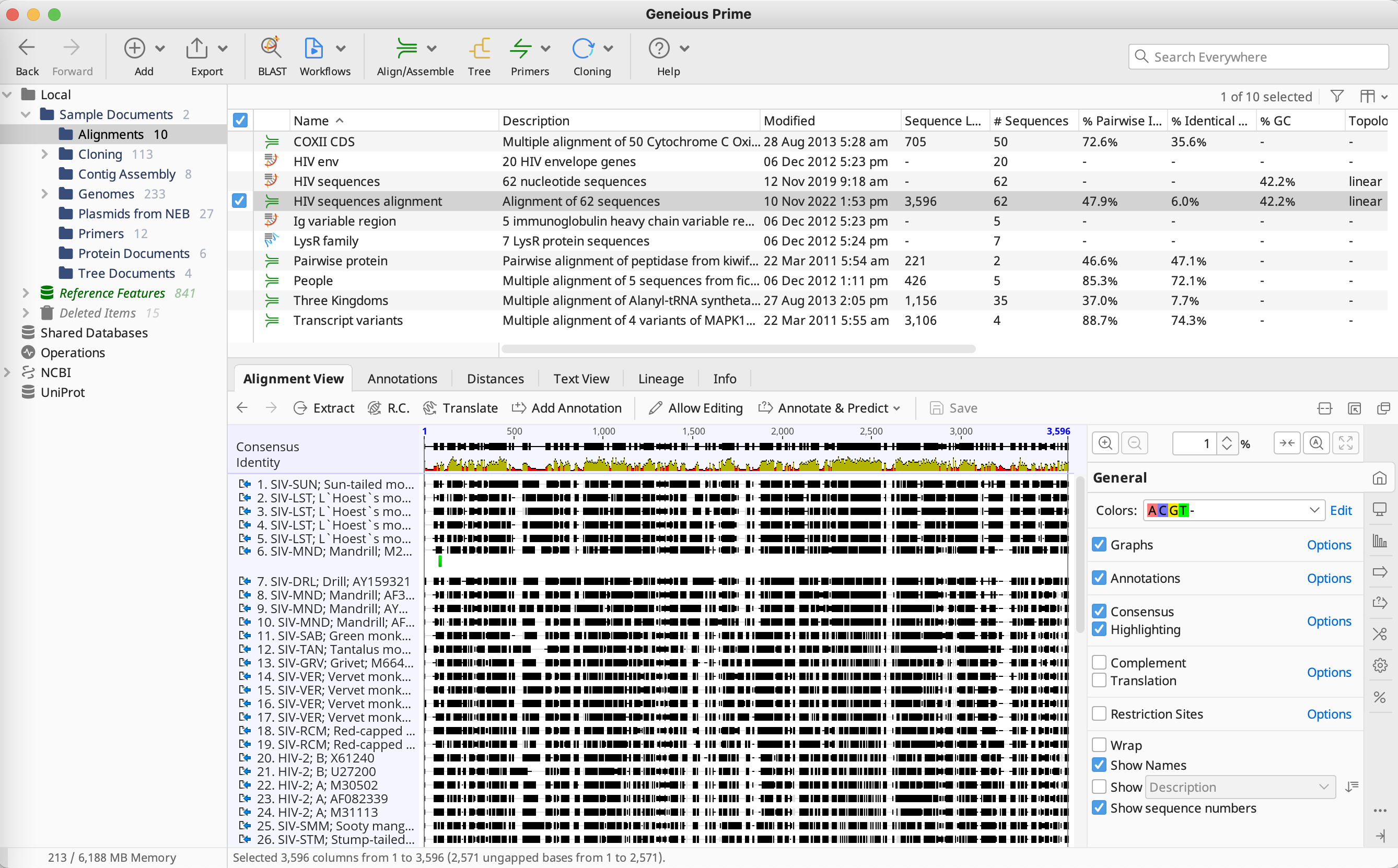 Alignment Viewでアラインされた塩基の配列を確認します。
Alignment Viewでアラインされた塩基の配列を確認します。
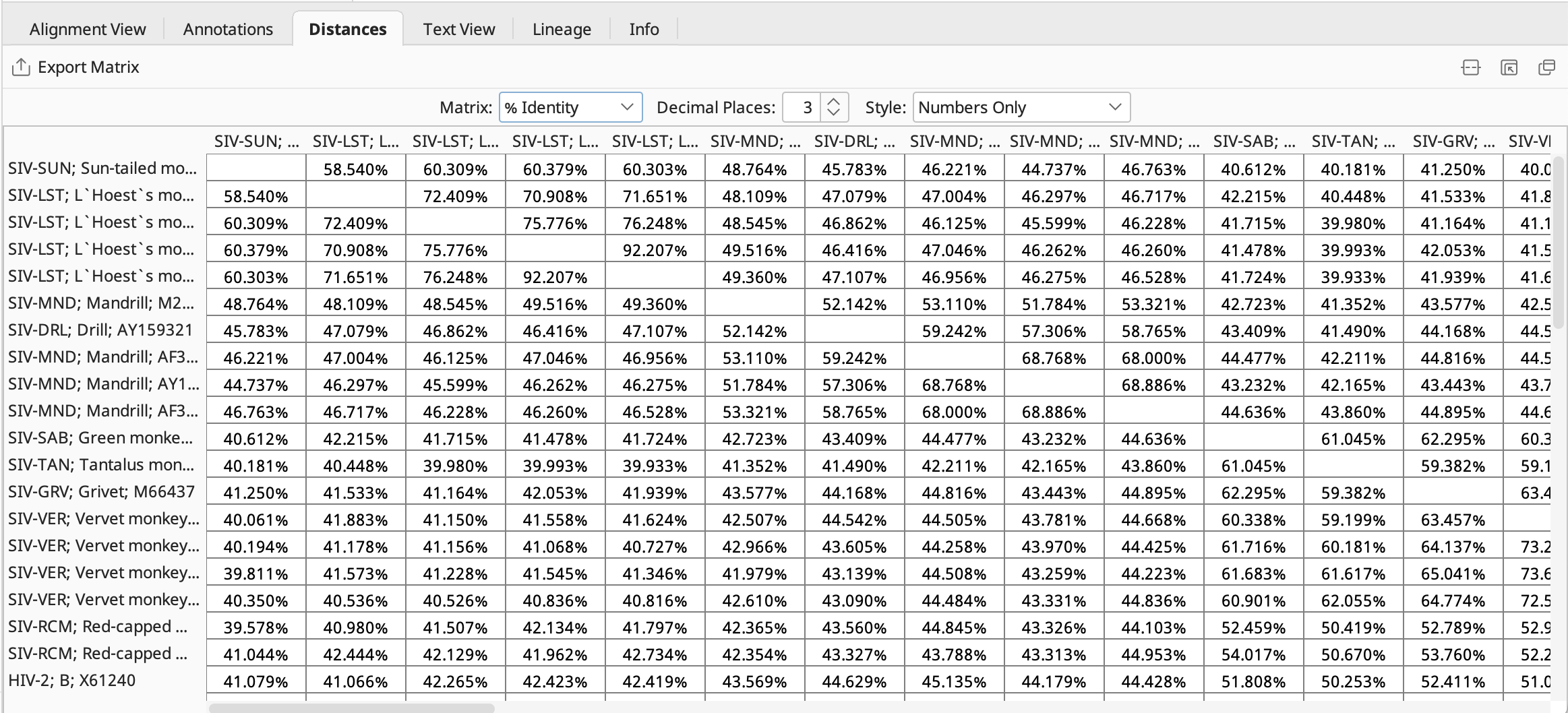
Distancesタブでは 、各配列間の相同性を確認することができます。Distances タブ の左上にある ![]() をクリックすると、表を CSV ファイルで出力できます。
をクリックすると、表を CSV ファイルで出力できます。
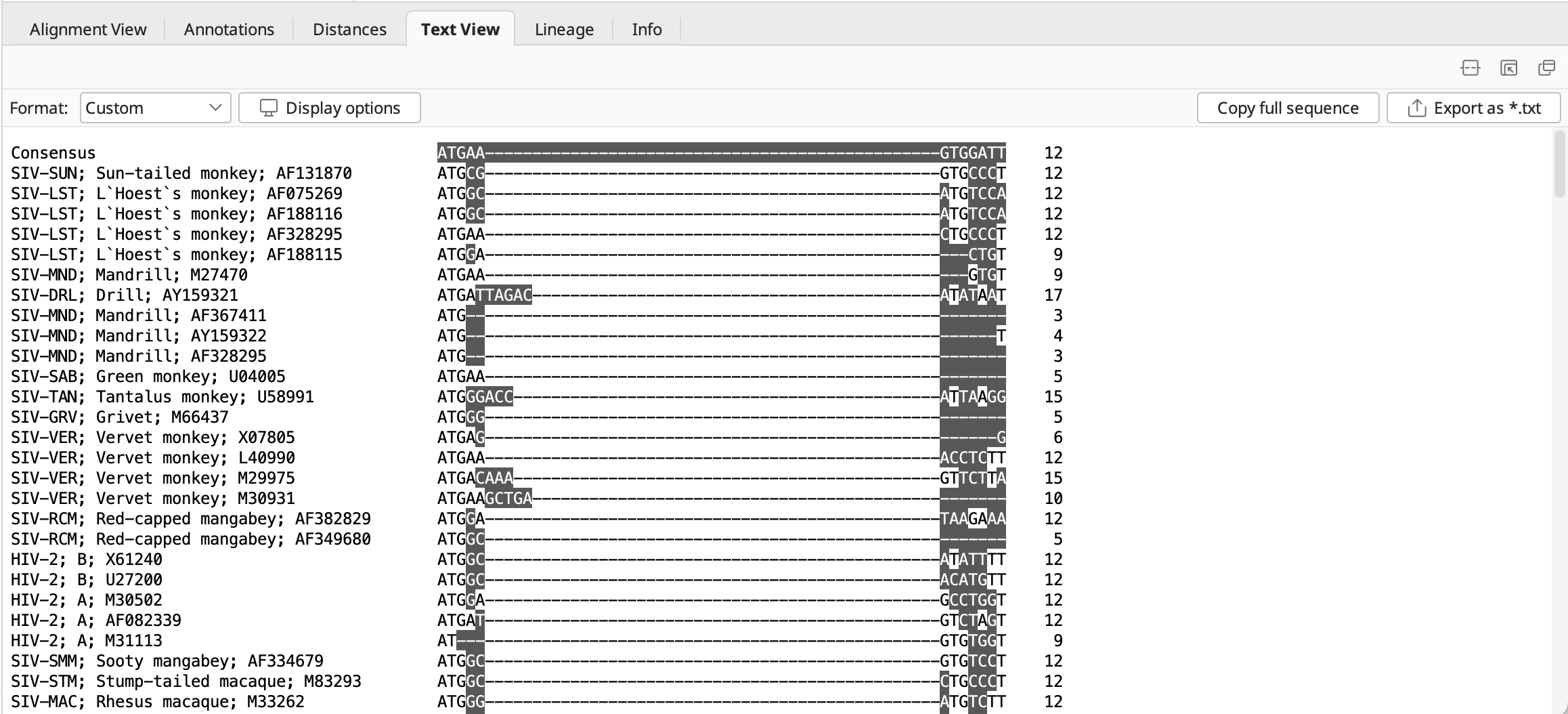
Text Viewタブでは、シンプルなアライメント結果を見ることができます。 ![]() をクリックすると、アライメント結果をテキストデータとして出力できます。
をクリックすると、アライメント結果をテキストデータとして出力できます。