制限酵素処理による挿入クローニング
制限酵素でインサートとベクターの切断・開環し、DNAリガーゼを使ってライゲーションを行うクローニングは最も一般的に行われている方法です。 SnapGeneでは、使用する制限酵素の選択からクローニング後のイメージまで、制限酵素とライゲーションによるクローニングのシミュレーションを簡単に行うことができます。またプロトコルに設計上の欠陥がある場合、そのエラーを発見して修正する機能も備えています。
このチュートリアルでは、一般的なクローニング法(制限酵素処理による挿入クローニング)をシミュレーションする方法をご紹介します。
1.【準備】DNA配列を入手する
本チュートリアルでは、A型インフルエンザウイルスの第8分節の配列と pcDNA3.1+/C-Myc を使用します。 下記ページよりA型インフルエンザウイルスの第8分節(アクセッション番号: NC_007370)の配列をコピーします。
www.ncbi.nlm.nih.gov
NC_007370.txt
1-1. ファイルの読み込み(Influenza A virus seg.8 配列)
SnapGene を起動して、右上の「新規」ボタンを選択し、「+ DNAファイル」をクリックします。
すでにSnapGeneを起動している場合は、メニューバーの 「ファイル」 →「 新しい DNA または RNA ファイル 」 から選択します。
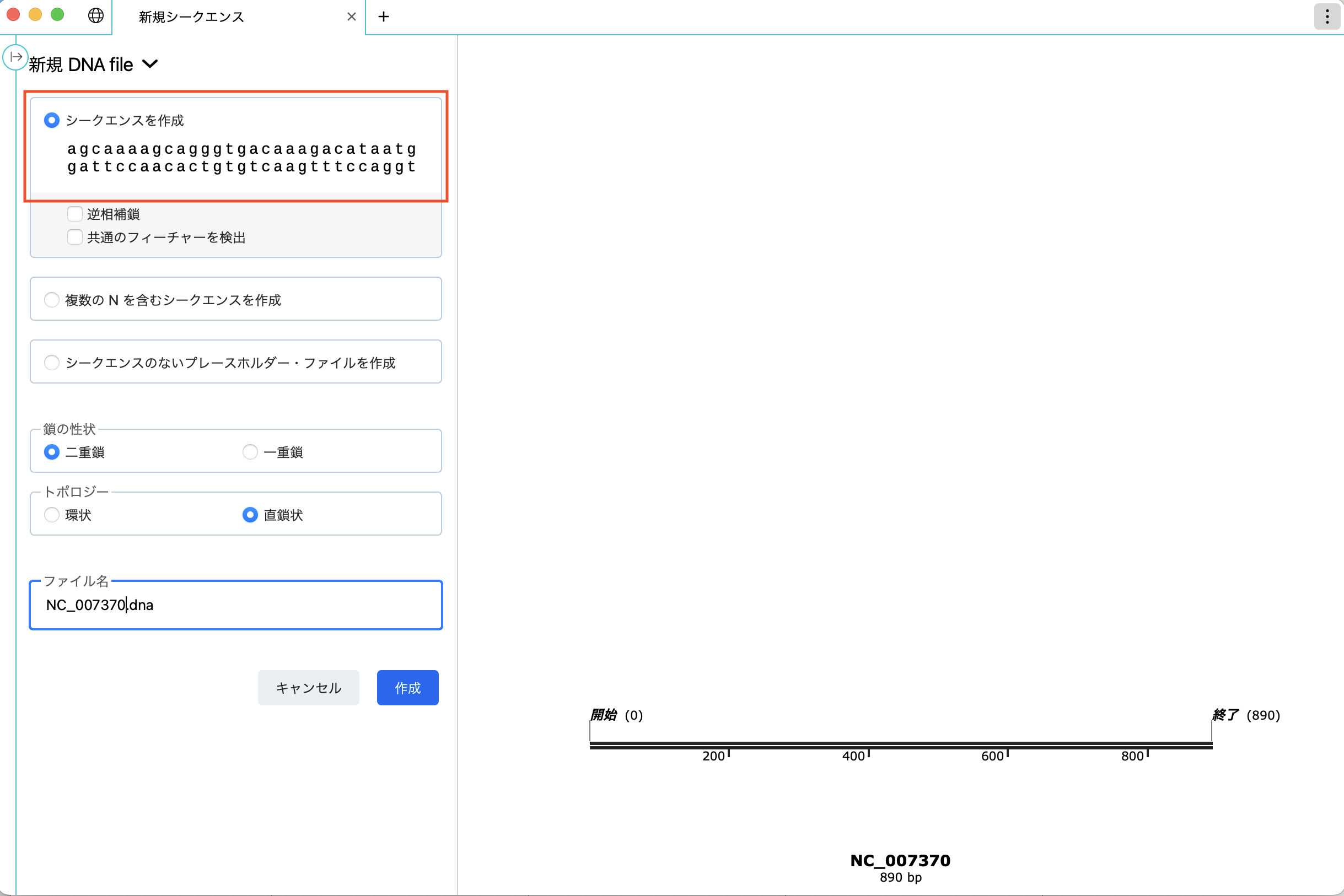
新規 DNA file フィールドにシークエンスをペーストし、「作成」ボタンをクリックします。
メニューバーの 「プライマー」 →「 プライマー追加 」を選択し、読み込んだ配列にプライマーを追加します。

以下のプライマーを追加しましょう。
EcoRI-IAV-NS1-Fw:CTCGAATTCATGGATTCCAACACTGTGTCAA
XhoI-IAV-NS1-Rv:GCGCTCGAGTTATCAAACTTTTGACCTAGCTGTTCT


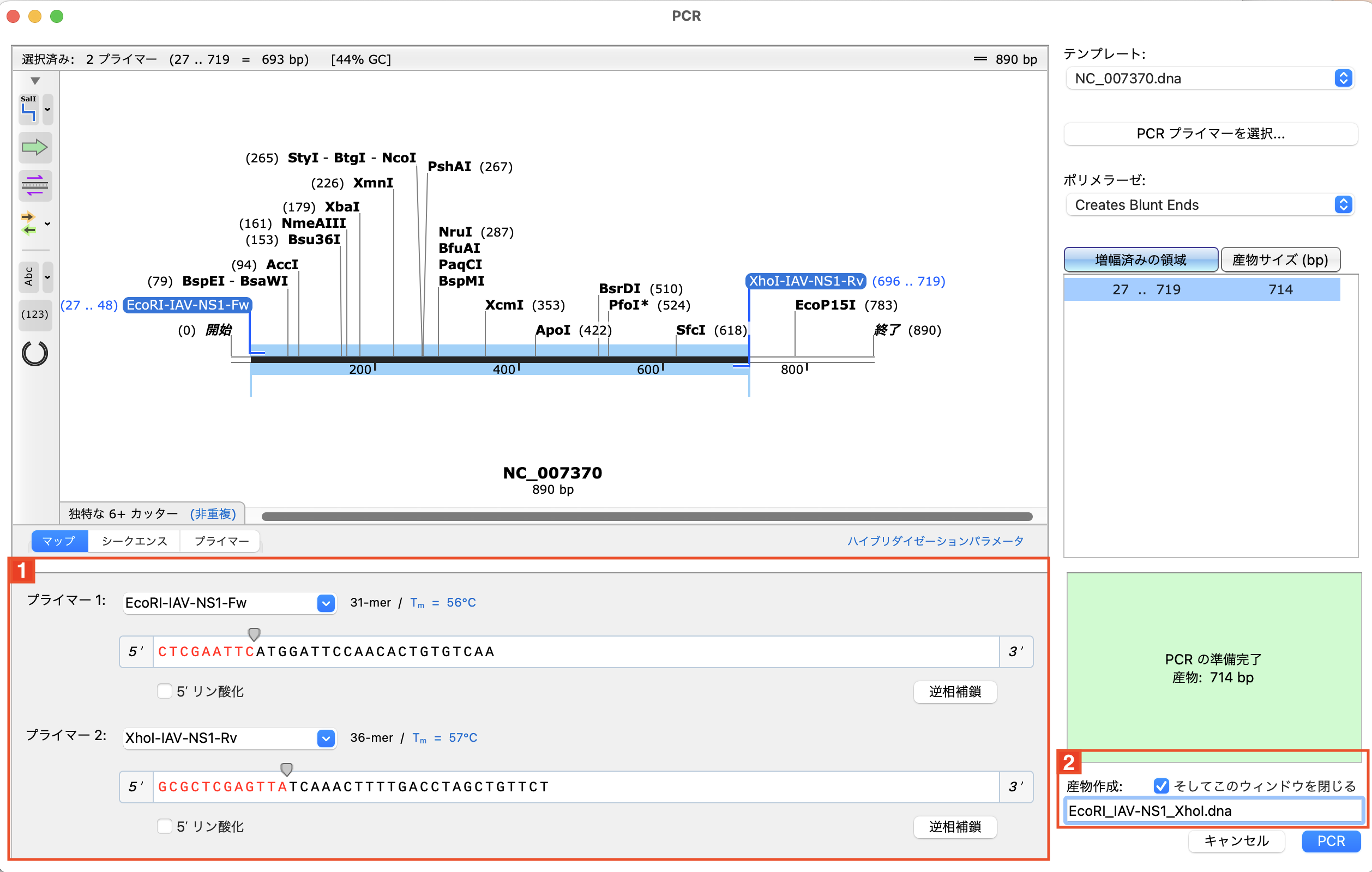
プライマーを追加後、PCRのシミュレーションを行います。メニューバーの「アクション」 →「PCR」を選択します。

1 先ほど作成したプライマーを、ドロップダウンメニューから選択します。2 ファイル名を入力し(ここでは 、EcoRI_IAV-NS1_XhoI.dna)、「PCR」ボタンをクリックし ます。
プライマーの作成およびPCRのシミュレーションの詳細に関しては、チュートリアルプライマーの作成とPCRのシミュレーションを参照してください。
1-2. ファイルの読み込み(pcDNA3.1+/C-Myc)
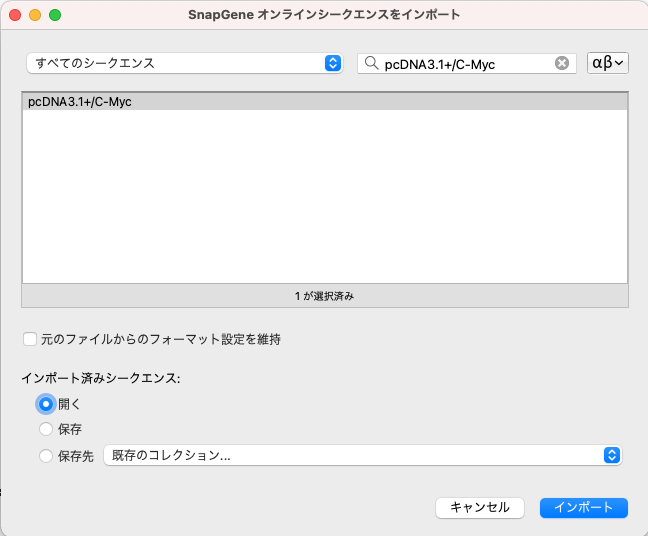
続いて、pcDNA3.1+/C-Myc を読み込みます。
ツールバーの「ファイル」→「インポート」→「SnapGeneオンラインシークエンス」を選びます。検索フィールドに “pcDNA3.1+/C-Myc” と入力し、ヒットした該当するオンラインシークエンスをインポートします。
「インポート」ボタンがアクティブ化されていない場合は、検索結果として表示された “pcDNA3.1+/C-Myc” をクリックして下さい。ボタンがアクティブ状態になります。

2. 制限酵素処理による挿入クローニング
本チュートリアルでは、pcDNA3.1+/C-Myc と PCRで増幅した EcoRI_IAV-NS1_XhoI を制限酵素(EcoRI と XhoI)で処理し、IAV-NS1をpcDNA3.1+/C-Myc にクローニングするという流れをシミュレーションします。
pcDNA3.1+/C-Myc の画面で、ツールバーの「アクション」から「制限酵素と挿入クローニング」「断片を挿入」へと進みます。

Ctrl または Shift を押しながら、制限酵素の EcoRI と XhoI を選びます。

次に「断片」タブ1 に切り替え、右側の「断片のソース」のドロップダウンリストから “EcoRI_IAV-NS1_XhoI.dna” 2 を選びます。

断片のソースを選択後、 制限酵素切を選択します。マップ上で Ctrl()/ shift()を押しながら、 EcoRI と XholI をクリックして選択します。

最後に「産物」タブに1 切り替えて、クローニング後のプラスミドマップを確認します。インサートは赤で、ベクターは黒で表示されています。 “pcDNA3.1-IAV-NS1-Myc.dna” 2 と名前を付けて、「クローン」ボタンをクリックします。

新しく開いた pcDNA3.1-IAV-NS1-Myc.dna の履歴ビューで、PCRからクローニングまでのシミュレーション履歴を見ることができます。





