バイオインフォマティクス解析ソフト Geneious
Geneious Prime概要 | ユーザー紹介 | 機能詳細 | システム条件 | 製品カタログ
Geneious Prime は、分子生物学実験シミュレーション機能、NGSデータの前処理、アセンブリ構築、マッピングツール、系統樹作成機能を備えたバイオインフォマティクス解析ソフトウェアです。直感的で使いやすい配列解析は、エラーとリスクを低減させ研究データの価値を最大限に引き出します。
Geneious Primeが選ばれる理由
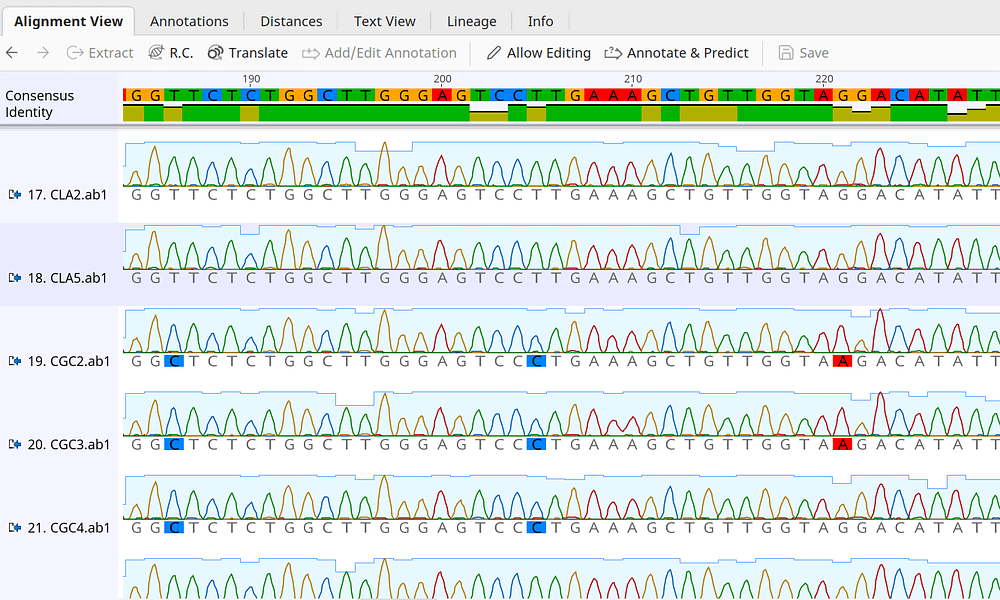
サンガーシークエンス法(ジデオキシ法)の解析
- シンプルかつ簡単な編集により、マッピング精度の向上と解析時間の短縮を実現します。
- 自動アノテーション機能により、遺伝子予測、モチーフ、アミノ酸翻訳、変異体検出が容易になります。
- マイクロサテライトの遺伝子型を特定し、対立遺伝子のテーブルを作成することが可能です。
次世代シークエンス(NGS)の解析
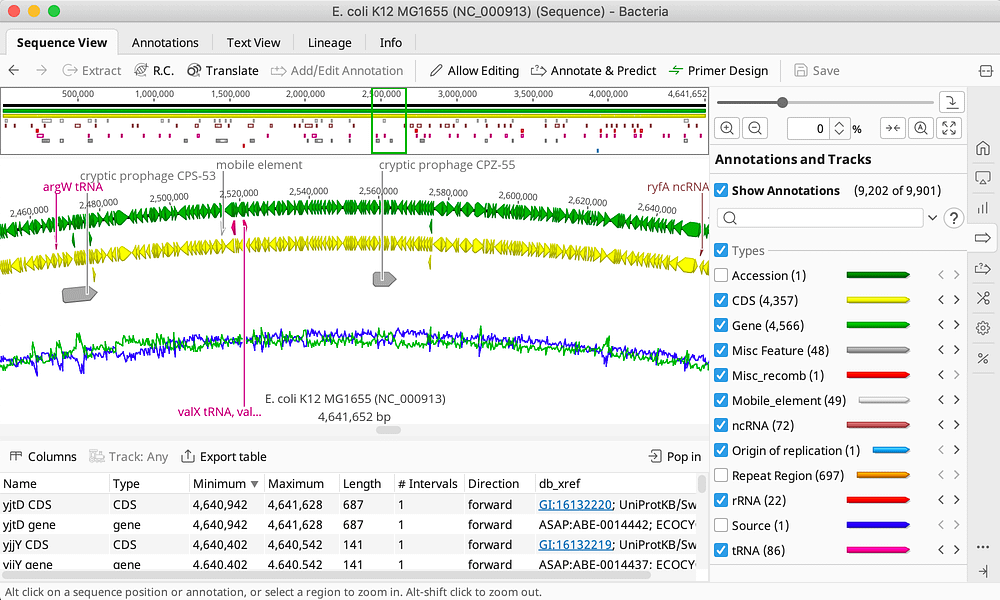
- アノテーション済みのゲノムやアセンブリを可視化します。シークエンスビューは、カスタマイズすることも可能です。
- 一塩基多型(SNP)の解析でアルゴリズムを選択できます。外部のパイプラインやプロバイダからのバリアントコールも利用可能です。
- RNA-Seqの発現解析は、プログラミング言語Rの専門知識を必要としません。
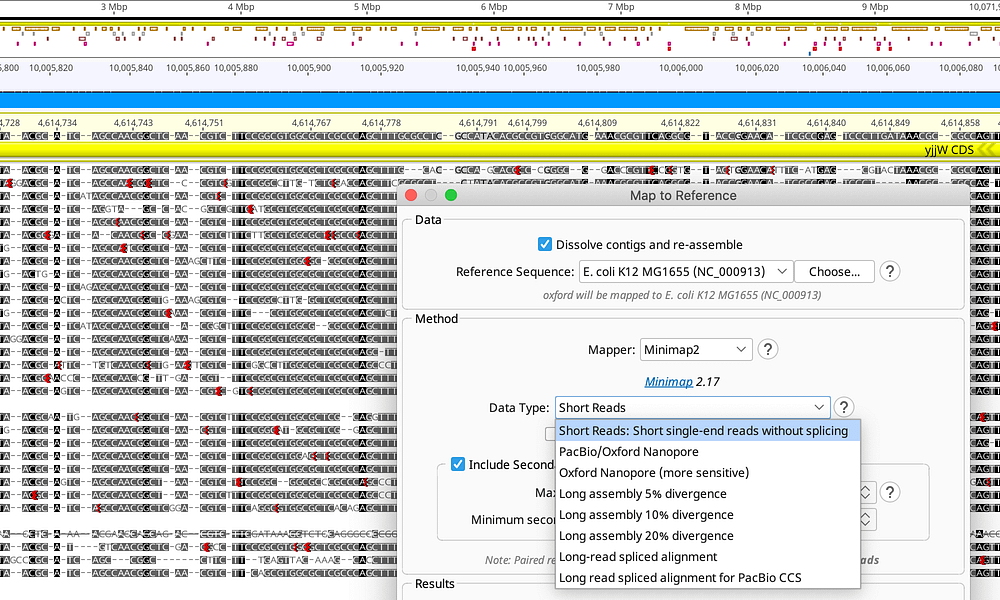
NGSデータの前処理、アセンブリ構築、マッピング
- クオリティの低いリードやコンタミネーションを除去する前処理ツールが豊富で、その後の解析に必要なデータ生成が可能です。
- Geneious独自のマッピングアルゴリズムで信頼性の高いリファレンスマッピングを提供します。
- de novoアセンブリで使用するデータは機器の種類を問いません。全ての次世代シークエンス(NGS)のデータを扱うことができます。また、比較のためのアライメントツールも提供しています。
アライメント
- Clustal Omega や MUSCLE などのサードパーティソフトウェアが用意されており、データセットのサイズを考慮してアルゴリズムを選択できます。
- MAFFT、MAUVE、LastZ をプラグインとして利用することが可能です。
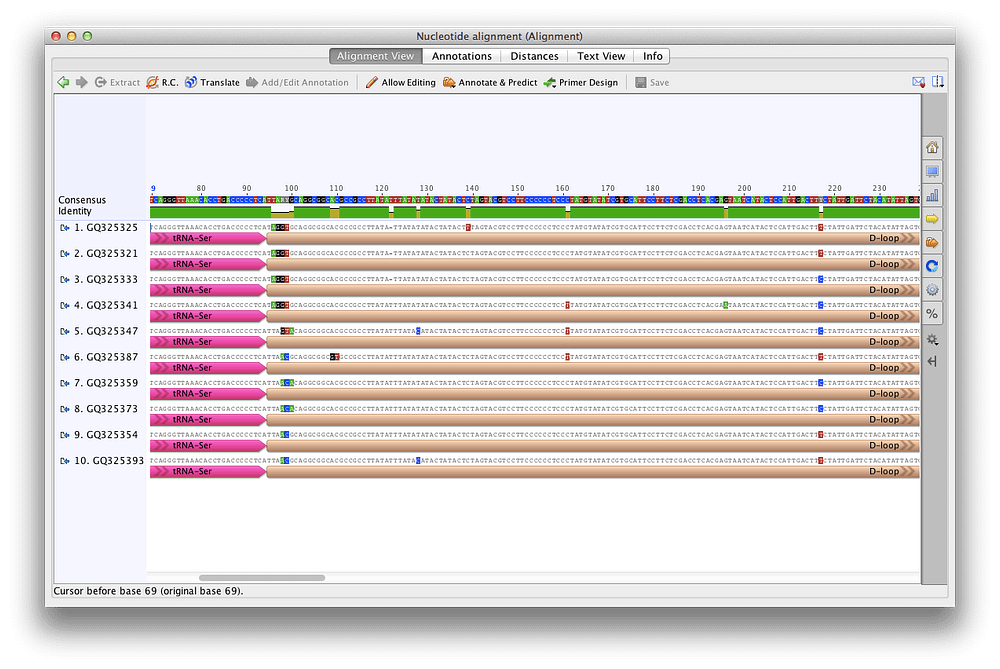
分子生物学実験のシミュレーション
- 制限酵素法、ゲートウェイ法、ゴールデンゲート法、インフュージョン法など、さまざまなクローニング法をシミュレーション可能です。
- PCRおよびシークエンス反応で使用するプライマーをデザインしシミュレーションすることができます。また、ユーザーが独自のプライマーデータベースを作成することができます。
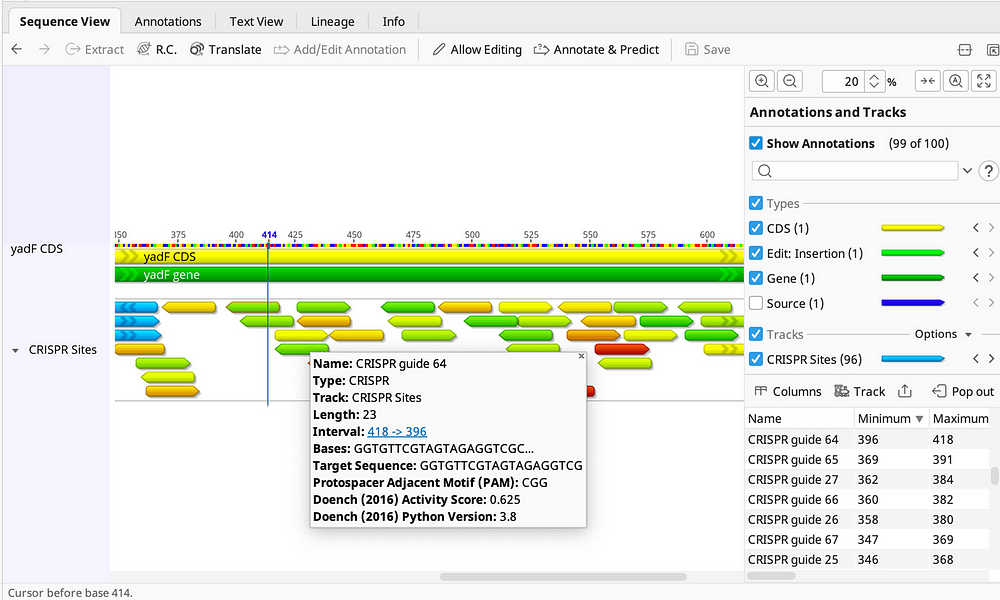
- CRISPRツールで、ターゲットの編集サイトの検索、gRNAの設計、編集結果の解析を行うことができます。
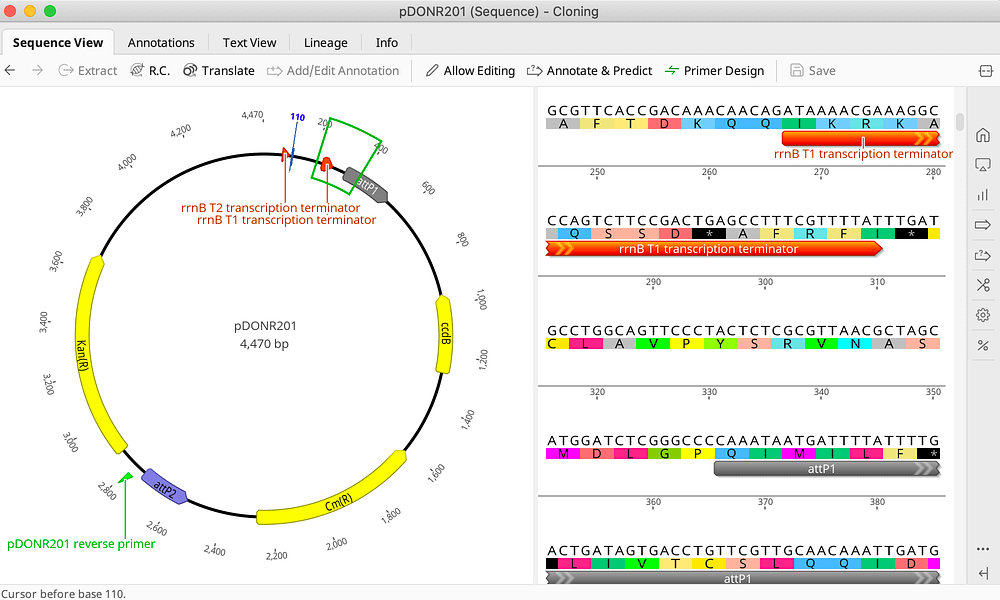
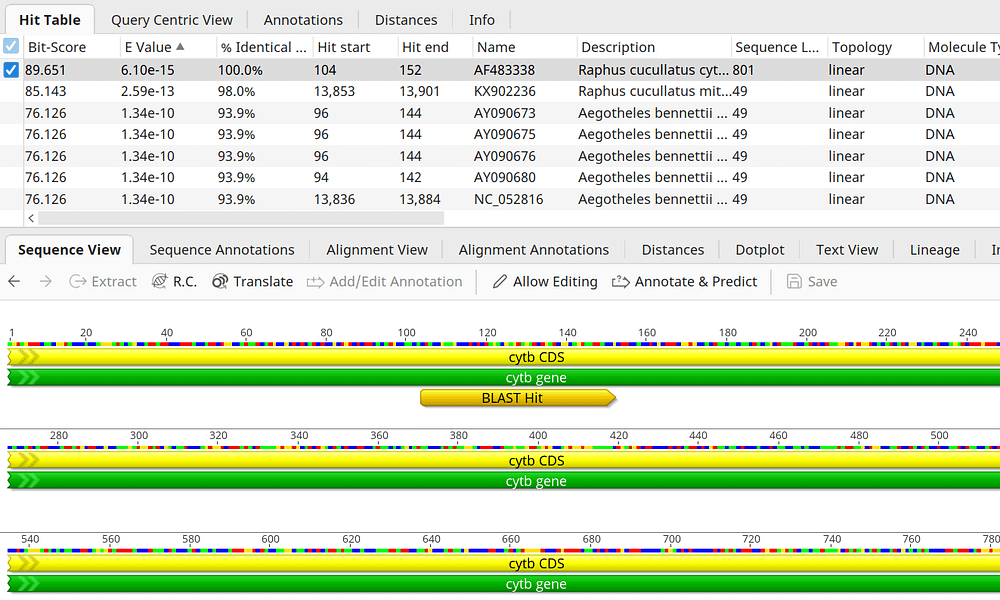
NCBIの利用とBLAST検索
- NCBIの主要なデータベースから、文献、DNA、タンパク質の配列情報を迅速に検索することが可能です。また、自動検索の設定をすれば、手動検索の代行をすることもできます。
- NCBIデータベースの配列に対して BLAST 検索を行い、その結果をGeneious Prime に反映させることができます。もちろん、ローカルのデータベースを設定してローカルで BLAST 検索を行うこともできます。
- ゲノム、フィーチャー、プライマートレースなどの配列をGenBank に登録することができます。
Geneious Prime 2024の新機能
Geneious Prime 2024.0では、クローニングバリデーション や De Novo Assembly 機能の向上、Map to Referenceアルゴリズムのアドバイザーの新機能が追加されました。
詳細はこちらから:Geneious Prime 新機能
研究を支える強力なバックボーン
 データマネジメント
データマネジメント
シームレスに統合された共有データベースは、プロセスの効率化とコラボレーションの向上を実現します。さらに、GenBank、SnapGene、FASTQを含む多くの一般的なファイルフォーマットをドラッグ&ドロップで簡単にインポート/エクスポートすることができます。
 カスタマイズ
カスタマイズ
Geneious Primeの機能を拡張するための各種プラグイン(アセンブリ、アラインメント、系統樹作成など)を用意しています。相互運用性の高い仕様となっていますので、既存のシステムとの統合や独自のカスタムアルゴリズムの追加も可能です。
 自動化
自動化
内蔵されたワークフローを使用することでエラーを減らすことができます。また、独自のワークフローを作成して、解析効率を高めることもできます。また、外部データベースの検索を自動化し、ゲノム・配列・タンパク質構造などの最新情報を継続的に入手することも可能です。
 イノベーション
イノベーション
Biomatters社の使命は、より早く・より効率的に科学研究を実施するためのプラットフォームを確立することです。Biomatters社では、Geneious Primeが正常に動作し、常に新しいリリースを提供できるように日々努力しています。
多くのユーザーに支持されています
Geneiousはバイオインフォマティクス研究を支援する強力なツールとして多くの研究者に利用されています。また Geneiousを使用した論文は、多くのジャーナルに広く掲載され引用されています。
Geneiousは、分子生物研究室にとって欠かせないツールですが、それだけではありません。設計、戦略やイノベーションのスピードを上げ、私達の研究を推進する原動力になっています。
"I find that Geneious is more than an invaluable tool for any modern molecular lab, it is an engine by itself that drives all our research and speeds up design, strategy and innovation."
Dr. Carlos Hidalgo-Grass,
Hadassah-Hebrew University
私達の研究室に Geneious は多大な貢献をしてくれています。ウェットとドライが混在するラボや、私たちのようなクローニングを多く行うラボにとって、非常に大きな価値を与えてくれると思います。
"Geneious has done a great deal for our lab, and I think it adds incredible value for labs with a mixture of wet and dry folks or those doing lots of cloning like ours."
Cole Trapnell,
Harvard University
Geneiousは、使いやすさとバイオインフォマティックの精妙さの完璧なコンビネーションです。
"Geneious is the perfect combination of ease-of-use and bioinformatic refinement."
Dr. Joshua Udall,
Plant and Wildlife Science Dept.
Brigham Young University
Geneious が使用された論文・ジャーナル
Geneious は分子生物学・細胞生物学・薬理学など生命科学分野における研究に利用され、データ解析結果が数多くの論文で引用されています。
- Application of next-generation sequencing for the high-resolution typing of MHC- B in Korean native chicken
-
"This study investigated five lines of indigenous Korean native chicken (KNC) and the Ogye breed using next-generation sequencing (NGS) data with Geneious Prime-based assembly and variant calling with the Genome Analysis Toolkit (GATK) best practices pipeline."
Ediriweera TK, Manjula P, Cho E, Kim M, Lee JH. Application of next-generation sequencing for the high-resolution typing of MHC-B in Korean native chicken. Front Genet. 2022 Oct 28;13:886376. doi: 10.3389/fgene.2022.886376. PMID: 36386795; PMCID: PMC9649677.
- Detection of clinically important β-lactamases by using PCR
-
"The functionality of proposed primers was verified using eight reference strains and 17 strains from our collection, which contained 29 different β-lactamase genes. PCR products of the test strains were confirmed by Sanger sequencing. Sequence analysis was performed using bioinformatics software Geneious."
Mlynarcik P, Dolejska M, Vagnerova I, Kutilová I, Kolar M. Detection of clinically important β-lactamases by using PCR. FEMS Microbiol Lett. 2021 Jun 16;368(11):fnab068. doi: 10.1093/femsle/fnab068. PMID: 34100944.
- Natural Wolbachia infection in field-collected Anopheles and other mosquito species from Malaysia
-
"Wolbachia sequences were aligned using Geneious 9.1.6 software, analyzed with BLAST, and the most similar sequences were downloaded."
Wong ML, Liew JWK, Wong WK, Pramasivan S, Mohamed Hassan N, Wan Sulaiman WY, Jeyaprakasam NK, Leong CS, Low VL, Vythilingam I. Natural Wolbachia infection in field-collected Anopheles and other mosquito species from Malaysia. Parasit Vectors. 2020 Aug 12;13(1):414. doi: 10.1186/s13071-020-04277-x. PMID: 32787974; PMCID: PMC7425011.
- RIM-binding protein couples synaptic vesicle recruitment to release sites
-
"The phylogenetic tree, multiple global sequence alignments (Blosum65), and editing (indications of deletion constructs, coiled-coil domain and interaction sites identified in cross-linking mass spectrometry) were performed in a Windows platform with the Geneious program version 9.1.8."
Astrid G. Petzoldt, Torsten W.B. Götz, Jan Heiner Driller, Janine Lützkendorf, Suneel Reddy-Alla, Tanja Matkovic-Rachid, Sunbin Liu, Elena Knoche, Sara Mertel, Vladimir Ugorets, Martin Lehmann, Niraja Ramesh, Christine Brigitte Beuschel, Benno Kuropka, Christian Freund, Ulrich Stelzl, Bernhard Loll, Fan Liu, Markus C. Wahl, Stephan J. Sigrist; RIM-binding protein couples synaptic vesicle recruitment to release sites. J Cell Biol 6 July 2020; 219 (7): e201902059. doi: https://doi.org/10.1083/jcb.201902059
- Identification of novel synaptonemal complex components in C. elegans
-
"We annotated the syntenic locus of each identified syp-5/6-related gene according to orthologous genes in C. elegans, and syp-5/6-related genes were named according to their shared syntenic location. SYP-5 and SYP-6 protein sequences were aligned using ClustalW (Larkin et al., 2007) implemented in Geneious Prime 2019.0.4. "
Matthew E. Hurlock, Ivana Čavka, Lisa E. Kursel, Jocelyn Haversat, Matthew Wooten, Zehra Nizami, Rashi Turniansky, Philipp Hoess, Jonas Ries, Joseph G. Gall, Ofer Rog, Simone Köhler, Yumi Kim; Identification of novel synaptonemal complex components in C. elegans. J Cell Biol 4 May 2020; 219 (5): e201910043. doi: https://doi.org/10.1083/jcb.201910043
- An ancient Sec10–formin fusion provides insights into actin-mediated regulation of exocytosis
-
"FH2 and SEC10 DNA sequences were translated into protein and aligned using MAFFT (Katoh and Standley, 2013), as implemented in Geneious (Kearse et al., 2012)."Peter A.C. van Gisbergen, Shu-Zon Wu, Mingqin Chang, Kelli A. Pattavina, Madelaine E. Bartlett, Magdalena Bezanilla; An ancient Sec10–formin fusion provides insights into actin-mediated regulation of exocytosis. J Cell Biol 5 March 2018; 217 (3): 945–957. doi: https://doi.org/10.1083/jcb.201705084
- Plastid Genome Evolution in the Subtribe Calypsoinae (Epidendroideae, Orchidaceae)
-
"The filtered paired reads were mapped to the plastome of Calanthe triplicata (NC_024544.1) using Geneious v. 10.1.2 to filter reads matching the reference genome."
Zhang-Hai Li, Yan Jiang, Xiao Ma, Jian-Wu Li, Jun-Bo Yang, Jian-Yong Wu, Xiao-Hua Jin, Plastid Genome Evolution in the Subtribe Calypsoinae (Epidendroideae, Orchidaceae), Genome Biology and Evolution, Volume 12, Issue 6, June 2020, Pages 867–870, https://doi.org/10.1093/gbe/evaa091
- Genomic characterization and phylogenetic analysis of SARS-COV-2 in Italy
-
"The results were mapped and aligned to the reference genome obtained from GISAID (https://www.gisaid.org/, accession ID: EPI_ISL_412973) using Geneious software, v. 9.1.5 (http://www.geneious.com)."
Zehender, G, Lai, A, Bergna, A, et al. Genomic characterization and phylogenetic analysis of SARS-COV-2 in Italy. J Med Virol. 2020; 92: 1637– 1640. https://doi.org/10.1002/jmv.25794
- SARS-CoV-2 and co-infections detection in nasopharyngeal throat swabs of COVID-19 patients by metagenomics
-
"Reference based mapping approach was applied to assemble SARS-CoV-2 genomes from the obtained sequences using Geneious 11.0.3 (Biomatters, Auckland, New Zealand)."
Van Tan, Le, et al. Journal of Infection, vol. 81, no. 2, 2020, pp. e175–77, http://doi.org/10.1016/j.jinf.2020.06.033.
- Extracellular chloride signals collagen IV network assembly during basement membrane formation
-
"Multiple sequence alignments were generated with GENEIOUS v.4.8.5 using the blosum62matrix."
Christopher F. Cummings, Vadim Pedchenko, Kyle L. Brown, Selene Colon, Mohamed Rafi, Celestial Jones-Paris, Elena Pokydeshava, Min Liu, Jose C. Pastor-Pareja, Cody Stothers, Isi A. Ero-Tolliver, A. Scott McCall, Roberto Vanacore, Gautam Bhave, Samuel Santoro, Timothy S. Blackwell, Roy Zent, Ambra Pozzi, Billy G. Hudson; Extracellular chloride signals collagen IV network assembly during basement membrane formation. J Cell Biol 23 May 2016; 213 (4): 479–494. doi: https://doi.org/10.1083/jcb.201510065
- Dynamics of in vivo ASC speck formation
-
"The software Geneious (version 6.1.7r; Biomatters Limited) was used for cloning-strategy design, sequencing data analysis, and sequence alignments."
Paola Kuri, Nicole L. Schieber, Thomas Thumberger, Joachim Wittbrodt, Yannick Schwab, Maria Leptin; Dynamics of in vivo ASC speck formation. J Cell Biol 4 September 2017; 216 (9): 2891–2909. doi: https://doi.org/10.1083/jcb.201703103
- UNC-45a promotes myosin folding and stress fiber assembly
-
"The Geneious (Biomatters Ltd.) analysis tool was used to construct sequence alignments in Fig. S4 D."
António J.M. Santos, Cristina Nogueira, Maria Ortega-Bellido, Vivek Malhotra; TANGO1 and Mia2/cTAGE5 (TALI) cooperate to export bulky pre-chylomicrons/VLDLs from the endoplasmic reticulum. J Cell Biol 9 May 2016; 213 (3): 343–354. doi: https://doi.org/10.1083/jcb.201603072
Geneious Prime機能詳細
Geneious Primeに搭載されている分子生物学・配列解析ツールの一覧です。
NGSデータの前処理
- Illumina・PacBio・NanoPoreのデータをインポート
- Trim・filter・demultiplex(single-end dataとpaired-end dataどちらでも可)
- オーバーラップするpaired readsをマージ
- De-duplicate(重複除外)
- エラーの補正と標準化
- キメラ領域(低オーバーラップ領域)を除去
マッピングアセンブリとde novoアセンブリ
- マッピングアセンブリとde novoアセンブリのアルゴリズム変更が簡単に行えます.
- ペアエンドリードおよびハイブリッドアセンブリーを含むサンガーおよびNGS(Illumina, PacBio and Oxford Nanoporeなど)のデータのアセンブリをサポート
- 微生物、プラスミドなどの環状ゲノムの編集
- MAUVE(コマンド入力の知識・技術不要の配列比較ツール)によるゲノム比較と仕上げ
- Minimap2などのマッパーを搭載し、さまざまなデータをアセンブル・マッピングすることが可能
- Geneiousオリジナル、Tadpole、Velvetなどに対応したDe novoアセンブリのアルゴリズムを搭載
変異検出や発現量解析
- 変異・SNPs検出
- ゲノムデータと同期した表形式の結果のフィルタリング
- マッピング後の発現量解析
- 主成分分析やvolcano plotの描画
シークエンス解析
- サンガー法のシークエンス編集や解析
- 塩基の修整やコンセンサス配列の検出
- 各種モチーフ、ORF、リピート配列のアノテーション編集
- 遺伝子や構造の予測
- 相同性検索によるリアルタイムのアノテーション
- 選択した任意の領域のみの翻訳、アノテーションや選択したフレームの翻訳
- 等電点(pI)、分子量、融点、アミノ酸組成などのグラフと統計情報の表示
アライメント
- 全ゲノムのアラインメントを含む、DNA・タンパク質のペアワイズおよび多重配列アラインメント
- MAFFT、Clustal Omega、MUSCLEなど幅広いアルゴリズムを搭載しているので、塩基・アミノ酸どちらもアラインメントが可能
- アライメントした塩基配列の翻訳とハイライト機能
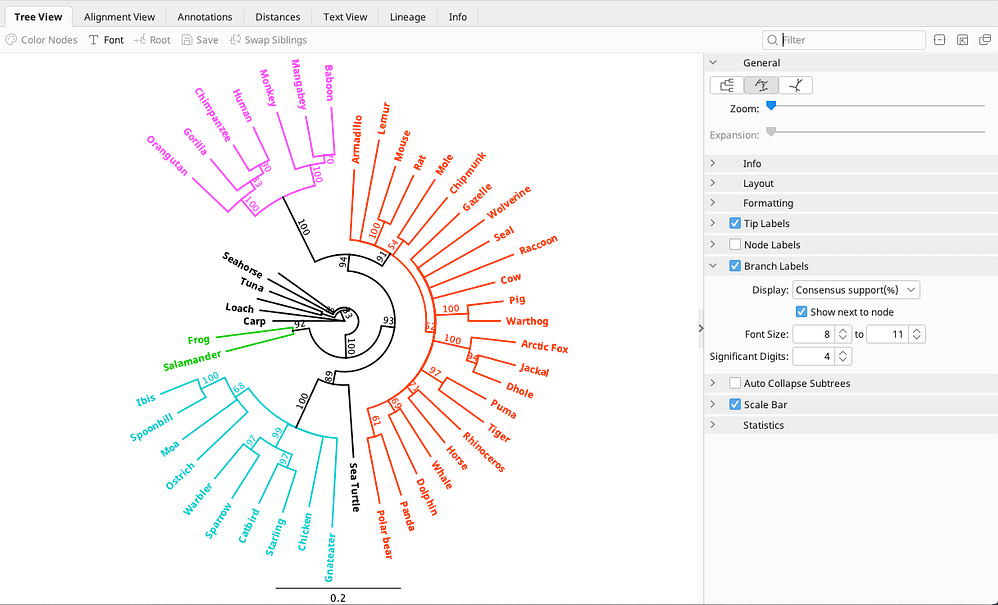
系統樹解析
- UPGMA、MrBayes、PAUP、PhyML、GARLI、RAxMLなど、さまざまなアルゴリズムを使って系統樹作成が可能
- 作成した系統樹の編集機能
- 各要素間の距離の可視化
- 論文等の掲載に有用な高品質の描画機能
マイクロサテライト解析
- ABIトレースファイルのインポート機能
- ピークの調整やトリミング機能
- ピークをアレルごとに分類
- アレル検出結果の表形式での出力
遺伝子クローニング
- プラスミドマップの可視化、任意ベクターの自動アノテーション、アノテーション付き配列のコピー&ペースト機能
- Golden Gate法(Type IIS制限酵素を用いた方法)および制限酵素法によるクローニングシミュレーション
- Gibson・GeneArt・In-Fusionなどを使ったシームレスクローニングのシミュレーション
- TOPOクローニング
- クローンニング操作の履歴追跡
- コドンオプティマイズと逆翻訳機能
- サイレント変異解析で導入可能な制限酵素配列を探す
- CRISPR-Cas9システムのシミュレーション
- PCR・制限酵素処理・ライゲーションのシミュレーション
プライマーの作成
- 任意のターゲット領域(または配列全体)に対して、PCR用・シークエンス用のプライマーやハイブリダイゼーションプローブを自動的にデザイン
- 配列データにプライマー情報の追加
- 通常のPCRおよびdegenerate PCRのプライマーデザイン
- プライマー配列の編集(追加・削除)も可能
- ターゲット配列上で他のアニーリング部位の有無をチェックするプライマー特異性試験
- 物理的特性、ヘアピン構造、プライマーダイマーなどのスクリーニング
- プライマー情報をFASTA、スプレッドシート、GenBankフォーマットにドラッグ&ドロップで追加可能
データマネジメントと互換性
- ドラッグ&ドロップでデータのインポートが可能
- 表計算ソフトやドキュメントファイル上のメタデータのインポート機能
- SAM、BAM、GFF、BED、VCFなどのあらゆる種類のファイルに対応
- フォルダベースでプロジェクト管理ができるので直感的な操作が可能
- データベースの共有機能
- 配列データだけでなく、メタデータの検索も可能
- 充実したエクスポート機能
検索とBLSAT機能
- NCBIのBLASTデータベースに直接アクセス可能
- 任意のデータベースを作成して、BLASTの実行も可能
- GenBankやUniProtなどのNCBI外のデータベースも検索可能
- GenBankにシークエンスをアップロード可能
- PubMedで文献検索も可能
- 検索機能をカスタマイズするアドバンス機能
ワークフローの自動化
- コマンドライン不要の自動化ワークフローの作成が可能
- 「参照配列にバリアントを適用」、「リードをマップしてSNPを検出」、「ランダムに配列をサンプリング」など20種類以上のワークフローをデフォルトで内蔵
- 既存ワークフローの解析設定も自由に編集可能
API開発
- 解析を拡張する種々のプラグインを無料で提供しています。
- 任意のアルゴリズムやデータベースを追加可能
- グラフィカルユーザインターフェース(GUI)で実行するコマンドラインプログラムを作成可能
Geneious Primeシステム条件
Geneious Primeのシステム条件は以下の通りです。インストール可能台数やライセンス許諾についてはFAQ:ライセンスについて をご参照下さい。
各OSシステム条件
Windows
Windows 7 ~ 11
macOS
Geneious Prime 2023: MacOS 10.14 以降
Geneious Prime 2020 ~ 2022: 10.11 以降
Geneious Prime 2019: MacOS 10.8 ~ 10.15*
Linux
Geneious R10 以降:Ubuntu Desktop LTS (18.04, 20.04).
Geneious R9 以前:Ubuntu Desktop LTS 16.04
OS の種類に関わらず、32 bit 版には対応しておりません。
HD空き容量 各OS共通
プロセッサ:x86 64-bit
メモリ容量:2 GB 以上
HD空き容量:2 GB 以上
ディスプレイ: 1024 x 768以上
Geneious製品カタログ
Geneious Primeのデジタルカタログをダウンロードいただけます。